遗传进化与表观基因组研究组

组长:段守富
电话:+86-010-64807527
电子邮件:duansf@im.ac.cn
个人简介
段守富,博士,研究员,博导,2018年毕业于中国科学院微生物研究所。博士学习期间和助理研究员工作期间,主要利用全基因组及表型谱数据对酿酒酵母群体遗传进行研究,揭示了中国是酿酒酵母的起源中心;同时结合分子遗传实验,阐明了马奶酒谱系适应性进化的分子机制。Current Biology期刊刊发了由美国加州大学伯克利分校 Rachel Brem教授撰写的专评文章,高度评价了相关研究成果。近年主要以酿酒酵母和哺乳动物细胞为模型,探究DNA复制过程中表观遗传信息传递的分子机制以及染色质重塑因子SMARCA4和CHD2如何影响胶质母细胞瘤的生长和发展等。近五年在国际期刊发表论文14篇,其中以第一作者(含共同)在Nature(2023)、Cancer Discovery(2022)、Nature Communications(2022,2018)、Science Advances(2022)、Current Biology(2019)等国际重要期刊发表6篇。曾获中国科学院优秀博士毕业论文以及中国科学院院长优秀奖等荣誉.
研究组研究方向
1. 酿酒酵母遗传多样性和适应性进化,微生物协同演化的分子机制
2. 表观遗传信息传递和细胞状态与命运的决定机制
研究组研究内容及意义
1. 酿酒酵母遗传多样性和适应性进化,微生物协同演化的分子机制
酿酒酵母(Saccharomyces cerevisiae)的野生和驯养种群在生态学、群体遗传学和群体基因组学等方面已经被广泛研究。然而,关于酿酒酵母驯化群体的起源方式,学术界仍存在争议。本课题组计划采用二代和三代测序技术获得基因组信息,旨在研究酿酒酵母遗传多样性,适应性进化的基本规律,以及物种间演化规律的保守性与差异性;阐明多物种协同演化的分子机制,特别是基因组结构变异和基因表达调控模式等。
2. 表观遗传信息传递和细胞状态与命运的决定机制
表观遗传信息主要存储在染色质上的化学修饰中,如DNA甲基化和组蛋白修饰等。在酿酒酵母中,表观遗传信息主要被亲本组蛋白传递和承载,以确保基因表达的继承、染色质结构的稳定和细胞特性的维持。虽然在酿酒酵母和哺乳动物细胞中,已经揭晓了两条主要的亲本组蛋白传递通路(Mcm2-Ctf4-Polα和Pol ε途径),但亲本组蛋白如何准确地被转移到复制的DNA链上以及亲本H3-H4二聚体上的修饰如何精确复制到新生(H3-H4)2四聚体上的分子机制仍是未解之谜。当前,酿酒酵母表观遗传修饰和群体遗传结构多集中于系统性的独立研究,对两者相关性研究极少,因此对二者之间相互作用的理解非常有限。本课题组将解析表观遗传信息传递的调控机制,并比较表观遗传和群体遗传变异对酿酒酵母群体进化的作用。
主要学习及工作经历
学习经历:
2012-09 至 2018-06, 中国,中国科学院微生物研究所, 微生物学, 博士
2008-09 至 2012-06, 中国,华南理工大学, 生物工程, 学士
工作经历:
2024-02 至 今, 中国,中国科学院微生物研究所, 研究员,博士生导师
2019-09 至 2024-02, 美国,哥伦比亚大学欧文医学中心, 博士后
2018-07 至 2019-09, 中国,中国科学院微生物研究所, 助理研究员
获奖情况
1. 2019年获“中国科学院优秀博士学位论文”奖
2. 2018年获“中国科学院院长优秀奖”
3. 2010年获“华南理工大学国家奖学金”
代表性论文(#共同一作,*通讯)
1. Duan SF, Han PJ, Wang QM, Liu WQ, Shi JY, Li K, Zhang XL, Bai FY*. The origin and adaptive evolution of domesticated populations of yeast from Far East Asia. Nature Communications. 2018, 9(1):2690.
2. Duan SF, Shi JY, Yin Q, Zhang RP, Han PJ, Wang QM, Bai FY*. Reverse evolution of a classic gene network in yeast offers a competitive advantage. Current Biology. 2019, 29(7):1126-1136.e5.
3. Serra-Cardona A, Duan SF#, Yu CH, Zhang ZG*. H3K4me3 recognition by the COMPASS complex facilitates the restoration of this histone mark following DNA replication. Science Advances. 2022, 8(18):eabm6246.
4. Xu XW, Duan SF#, Hua X, Li ZM, He R, Zhang ZG*. Stable inheritance of H3.3-containing nucleosomes during mitotic cell divisions. Nature Communications. 2022, 13(1):3533.
5. Mo Y, Duan SF#, Zhang X, Hua X, Zhou H, Wei HJ, Watanabe J, McQuillan N, Su ZY, Gu W, Wu CC, Vakoc CR, Hashizume R, Chang K, Zhang ZG*. Epigenome programming by H3.3K27M mutation creates a dependence of pediatric glioma on SMARCA4. Cancer Discovery. 2022, 12(12):2906-29.
6. Yang X, Duan SF#, Li ZM, Wang Z, Kon N, Zhang ZG*, Gu W*. Protocol of CRISPR-Cas9 knockout screens for identifying ferroptosis regulators. STAR Protocols. 2023, 4(4):102762.
7. Li Z, Duan SF#, Hua X, Xu XW, Li YL, Menolfi D, Zhou H, Lu C, Zha S, Goff SP, Zhang ZG*. Asymmetric distribution of H3K9me3 at replicating DNA strands silences L1 retrotransposons during S phase. Nature. 2023, 623:643-651.
其他合作文章
1. Han DY, Han PJ, Rumbold K, Koricha AD, Duan SF, Song L, Shi JY, Li K, Wang QM, Bai FY*. Adaptive gene content and allele distribution variations in the wild and domesticated populations of Saccharomyces cerevisiae. Frontiers in Microbiology. 2021, 12:631250.
2. Song L, Shi JY, Duan SF, Han DY, Li K, Zhang RP, He PY, Han PJ, Wang QM, Bai FY*. Improved redox homeostasis owing to the up-regulation of one-carbon metabolism and related pathways is crucial for yeast heterosis at high temperature. Genome Research. 2021, 31(4):622-634.
3. Yang X, Wang X, Li Z, Duan SF, Li H, Jin J, Zhang Z, Gu W*. An unexpected role for Dicer as a reader of the unacetylated DNA binding domain of p53 in transcriptional regulation. Science Advances. 2021, 7(44):eabi6684.
4. Evrin C, Serra-Cardona A, Duan SF, Mukherjee PP, Zhang Z*, Labib KPM*. Spt5 histone binding activity preserves chromatin during transcription by RNA polymerase II. EMBO Journal. 2022, 41(5):e109783.
5. Bai FY*, Han DY, Duan SF, Wang QM. The ecology and evolution of the baker’s yeast Saccharomyces cerevisiae. Genes. 2022, 13(2):230.
6. He PY, Shao XQ, Duan SF, Han DY, Li, K, Shi JY, Zhang RP, Han PJ, Wang QM, and Bai FY*. Highly diverged lineages of Saccharomyces paradoxus in temperate to subtropical climate zones in China. Yeast. 2022, 39, 69-82.
7. Su ZY, Kon N, Yi JJ, Zhao HQ, Zhang WW, Tang QS, Li H, Kobayashi H, Li ZM, Duan SF, Liu YQ, Olive KP, Zhang ZG, Honig B, Manfredi JJ, Rustgi AK, Gu W*. Specific regulation of BACH1 by the hotspot mutant p53(R175H) reveals a distinct gain-of-function mechanism. Nature Cancer. 2023, 4:564-581.
8. Yang X, Wang Z, Zandkarimi F, Liu YQ, Duan SF, Li ZM, Kon N, Zhang ZG, Jiang XJ, Stockwell BR, Gu W*. Regulation of VKORC1L1 is critical for p53-mediated tumor suppression through vitamin K metabolism. Cell Metabolism. 2023,35(8):1474-1490.e8.
9. Yang X, Wang Z, Samovich SN, Kapralov AA, Amoscato AA, Tyurin VA, Dar HH, Li ZM, Duan SF, Kon N, Chen D, Tycko B, Zhang ZG, Jiang XJ, Bayir H, Stockwell BR, Kagan VE, Gu W*. PHLDA2-mediated phosphatidic acid peroxidation triggers a distinct ferroptotic response during tumor suppression. Cell Metabolism.2024.
研究中代表性图片
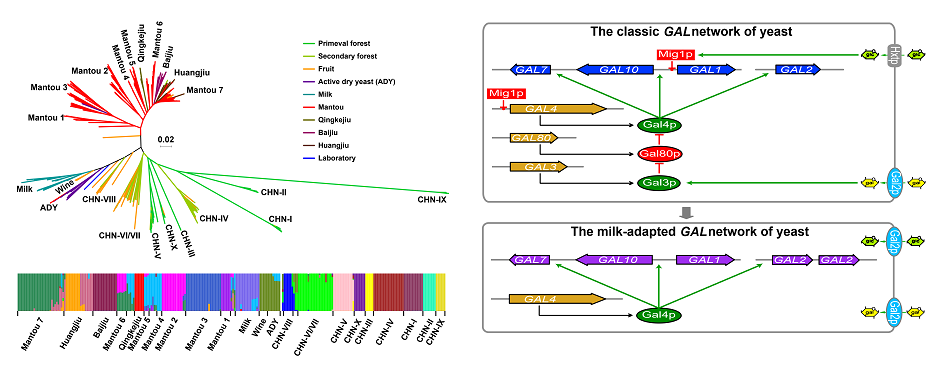
Duan et al., Nature Communications, 2018 Duan et al., Current Biology, 2019
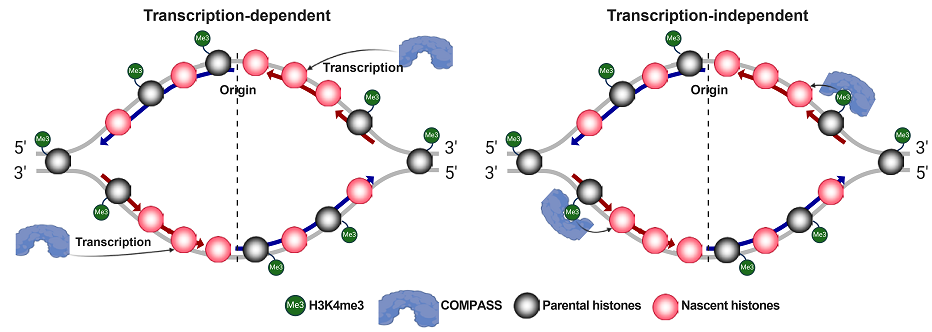
Serra-Cardona and Duan et al., Science Advances, 2022
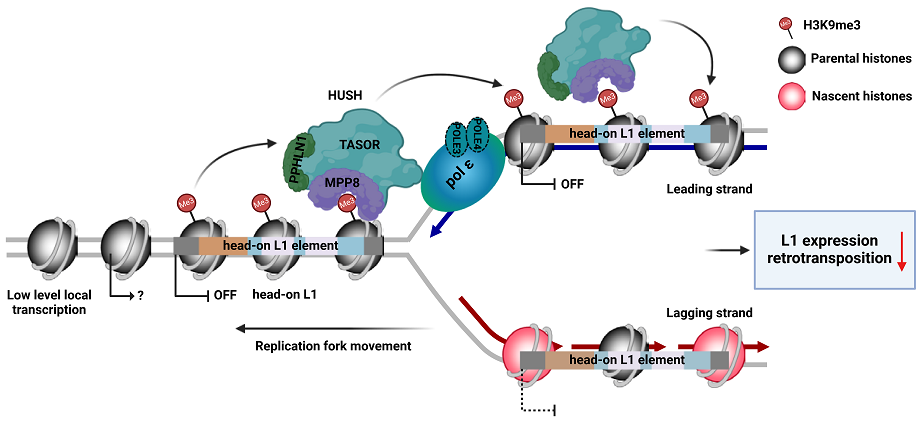
Li and Duan et al., Nature, 2023



 京公网安备 11010502044263号
京公网安备 11010502044263号